Как восстановить страницу в Контакте после удаления
Рассмотрим, как восстановить доступ к странице в Контакте после удаления без потери личных данных, фото, музыки и сохраненных переписок.
Пользователям социальной сети Вконтакте доступна функция удаления своей личной страницы.
Само удаление подразумевает два направления:
- Временная деактивация. Сразу после активации функции вы можете полностью закрыть доступ к страничке или оставить её для публичного просмотра. Для восстановления профиля достаточно повторно авторизоваться на сайте;
- Полное удаление аккаунта. Если на протяжении семи месяцев пользователь не восстановил страницу, она удаляется из базы данных сайта и её восстановление больше невозможно.
Также, аккаунт может быть заблокирован администрацией за нарушение правил сайта (рассылка спама, экстремистская деятельность, мошенничество, жалобы других пользователей).
Cодержание:
Способ 1 – Стандартное возобновление доступа
В большинстве случаев пользователи самостоятельно удаляют свои страницы. Восстановить её можно в течении 7-ми месяцев.
Для этого выполните такие действия:
- Зайдите на VK.com;
- Введите свой логин и пароль и нажмите Enter;
- Далее появится окно с информацией о том, что аккаунт был удален. Вверху вы увидите сообщение с указанием точной даты, когда профиль уже нельзя будет вернуть;
- Нажмите на поле «Восстановить». После этого вы попадете в окно своего профиля и сможете продолжить использовать социальную сеть.
Рис.2 – деактивированная страничка
Заметим, что после возобновления доступа сохраняются абсолютно все ваши данные: фотографии, отдельные альбомы, музыка, диалоги, записи и списки сообществ.
В любой момент вы можете повторно удалить страницу через настройки.
Способ 2 – Если логин и пароль утерян
Первый способ подразумевает простое возобновление аккаунта, при котором нужно обязательно знать логин или пароль доступа.
Если таких данных у вас нет, либо ваш пароль почему-то не подходит, придется восстанавливать профиль другим методом.
Потеря пароля
К каждой страничке социальной сети пользователи привязывают номер мобильного. Это обязательное требование при регистрации.
В дальнейшем этот номер можно использовать вместо логина на этапе авторизации.
Вспомните, к какому номеру привязан ваш аккаунт.
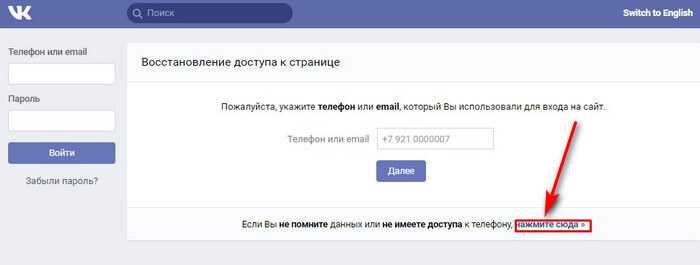
Затем зайдите на сайт социальной сети и внизу возле формы ввода логина и пароля нажмите на «Забыли пароль?»:
Затем в новом окне нужно ввести номер телефона в международном формате.
После этого сайт попросит ввести капчу (символы с картинки).
На следующем этапе сайт задаст вам несколько вопросов, чтобы удостовериться в том, что именно вы являетесь владелец странички.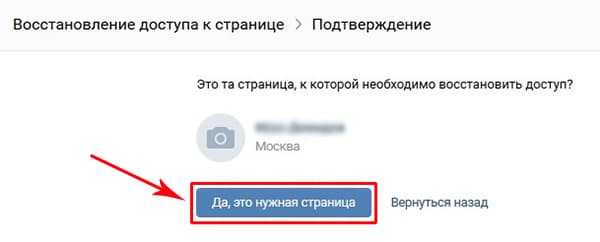
Введите фамилию, которая указана в профиле и нажмите «Далее»:
Убедитесь в том, что найденная системой страница действительно ваша. Часто сайт предлагает ошибочные профили.
Это происходит из-за большого количества пользователей с одинаковыми именами.
Нажмите на «Да, это я» или вернитесь назад и проверьте правильность ввода мобильного.
В появившемся окне вы увидите ваш номер телефона и кнопку «Получить код».
Нажмите на неё. Через несколько секунд на телефон придет СМС-сообщение от сайта, в котором будет указан набор цифр.
Его вы должны ввести в следующем окне.
Если код долго не приходит, повторите попытку отправки сообщения, проверьте подключение к сотовой сети на телефоне или правильность написания номера.
Рис.7 – отправка СМС с кодом
Введите полученные цифры в указанное поле и нажмите Enter:
Рис.8 – ввод кода
На заключительном этапе восстановления сайт попросит придумать новый пароль.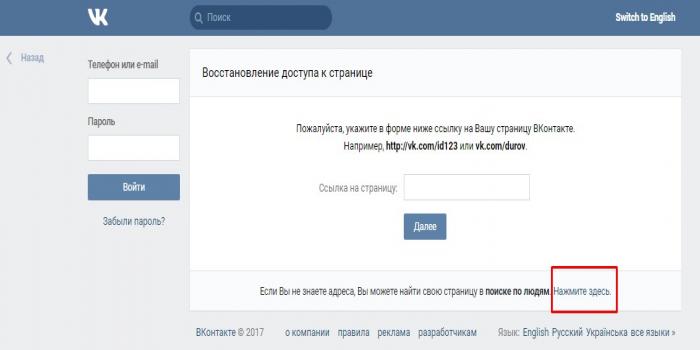 После этого вы можете вернуться к использованию личного аккаунта.
После этого вы можете вернуться к использованию личного аккаунта.
Нет логина и пароля
Бывают случаи, когда у пользователя нет нужного пароля и доступ к мобильному номеру тоже утерян.
Решить проблему можно следующим образом:
- В окне стартовой страницы нажмите на поле «Забыл пароль», как это показано на Рисунке 3;
- Затем кликните на поле «Если не помните данных»;
Рис.9 – возобновление доступа без логина и пароля
- В новом окне скопируйте ссылку на свою страничку или воспользуйтесь поиском по людям;
- Далее введите любой доступный вам номер телефона и нажмите «Далее»;
- Теперь нужно сфотографироваться на фоне монитора с этой заявкой. Важно чтобы ваше лицо и надписи на экране устройства были отчетливо видны. Рекомендуется делать снимок при дневном освещении.
- На следующем этапе загрузите цветные сканкопии первых двух страниц своего паспорта.
 Это необходимо для того, чтобы администрация смогла вас идентифицировать и вернуть доступ к аккаунту.
Это необходимо для того, чтобы администрация смогла вас идентифицировать и вернуть доступ к аккаунту.
- Добавьте личный комментарий. Опишите, почему доступ к странице был потерян. Указаны ли в профиле реальные данные о вас;
- Нажмите на «Подать заявку».
Далее потребуется еще раз подтвердить создание заявки кодом из СМС-сообщения.
Ожидайте рассмотрения ваших данных администрацией. Как правило, это занимает 1-7 дней.
Если спустя это время вам так и не изменили привязанный к аккаунту номер, это означает, что заявка не была одобрена.
Почему отказывают в выполнении заявки?
- Фото слишком низкого качества или их отсутствие;
- Указанные вами данные совсем не совпадают с теми, которые указаны на странице. Владельцам фейковых аккаунтов практически невозможно восстановить доступ. Если вашу заявку всё-таки одобрили, имя страницы будет изменено на то, которое указано в предоставленных документах;
- Указанный новый номер телефона уже привязан к другой странице во Вконтакте.

Способ 3 – Аккаунт заблокирован администрацией сайта
Если с вашего аккаунта было отправлено большое количество спама, его могут автоматически заблокировать.
Под спамом подразумевается массовая рассылка однотипных комментариев и сообщений, призывы к экстремистским действиям.
Также, на вас могло пожаловаться большое количество пользователей социальной сети.
После авторизации вы увидите не главную страницу с вашим фото, а такое окно:
В окне указывается причина блокировки и её период. Доступ к аккаунту может быть закрыт на сутки или навсегда.
Как правило, если вы столкнулись с такой проблемой впервые, вас заблочат всего на день в качестве предупреждения. Повторное нарушение правил сайта ведет к необратимой блокировке.
Что нужно делать?
Пользователь никак не может ускорить процесс возобновления доступа в случае принудительной блокировки. Рекомендуем зайти на сайт через сутки и обязательно изменить пароль.
Рекомендуем зайти на сайт через сутки и обязательно изменить пароль.
Если спам рассылали не вы, вероятно, вас взломали. Кто-то вошел под вашим именем и нарушил правила сайта.
Никогда не передавайте другим людям свой логин или пароль.
Также, старайтесь подобрать сложное кодовое слово, чтобы различные боты и брутфорсеры не смогли получить доступ к аккаунту с помощью автоподбора паролей.
Также, не оставляйте свои логины и пароли на других сайтах, в закладках электронной почты, личных сообщениях с другими пользователями.
к содержанию ↑Альтернативный вариант восстановления
Всегда можно обратиться напрямую в службу поддержки сайта.
Они помогут решить проблему блокировки или разобраться с трудностями в процессе восстановления.
Перейдите на страницу и выберите интересующую вас тему обращения.
В новом окне опишите ситуацию и дождитесь ответа администратора. Как правило, это занимает не более 1 часа.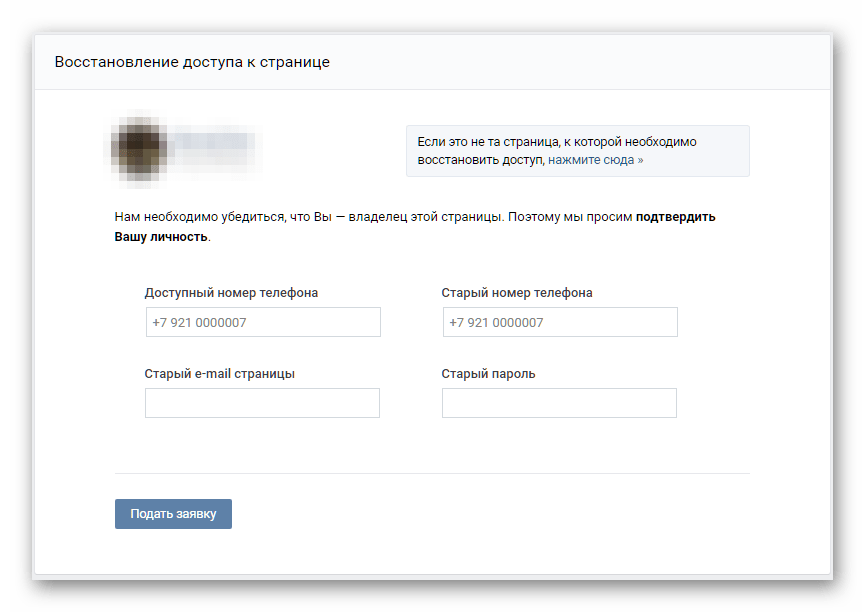
Тематические видеоролики:
Нацеленная интеграция ДНК в клетки человека без разрывов двухцепочечных фрагментов с использованием CRISPR-ассоциированных транспозаз
Pickar-Oliver, A. & Gersbach, C. A. Следующее поколение технологий и приложений CRISPR-Cas. Нац. Преподобный Мол. Клеточная биол. 20 , 490–507 (2019).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Нотт, Г. Дж. и Дудна, Дж. А. CRISPR-Cas определяет будущее генной инженерии. Наука 361 , 866–869 (2018).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Анзалон А.В., Коблан Л.В. и Лю Д.Р. Редактирование генома с помощью нуклеаз CRISPR-Cas, базовых редакторов, транспозаз и основных редакторов. Нац. Биотехнолог. 38 , 824–844 (2020).
Артикул КАС пабмед Google Scholar
Маруяма Т. и др. Ингибирование негомологичного соединения концов повышает эффективность CRISPR/Cas9-опосредованного точного редактирования генома. Природа 33 , 538–542 (2015).
КАС Google Scholar
Накаде, С. и др. Опосредованная микрогомологией интеграция донорской ДНК, зависящая от соединения концов, в клетках и животных с использованием TALEN и CRISPR/Cas9.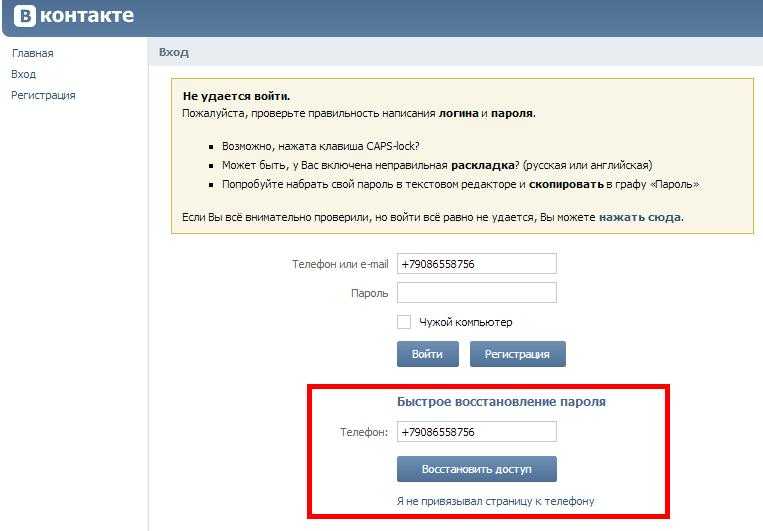
Артикул КАС пабмед Google Scholar
Чу, В. Т. и др. Повышение эффективности направленной по гомологии репарации точного редактирования генов, индуцированного CRISPR-Cas9, в клетках млекопитающих. Нац. Биотехнолог. 33 , 543–548 (2015).
Артикул КАС пабмед Google Scholar
Yeh, C.D., Richardson, C.D. & Corn, J.E. Достижения в редактировании генома посредством контроля путей репарации ДНК. Нац. Клеточная биол. 21 , 1468–1478 (2019).
Артикул КАС пабмед Google Scholar
Хейер В.-Д., Эмесн К.Т. и Лю Дж. Регуляция гомологичной рекомбинации у эукариот. год. Преподобный Жене. 44 , 113–139 (2010).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Moynahan, M.E. & Jasin, M. Митотическая гомологичная рекомбинация поддерживает стабильность генома и подавляет онкогенез. Нац. Преподобный Мол. Клеточная биол. 11 , 196–207 (2010).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Лин, С., Стаал, Б. Т., Алла, Р. К. и Дудна, Дж. А. Усовершенствованная гомологическая инженерия генома человека путем контролируемого времени доставки CRISPR/Cas9. eLife 3 , e04766 (2014).
Артикул пабмед ПабМед Центральный Google Scholar
Zuccaro, M.V. et al. Аллель-специфическое удаление хромосом после расщепления Cas9 у эмбрионов человека. Сотовый 183 , 1650–1654 (2020).
Артикул КАС пабмед Google Scholar
Adikusuma, F.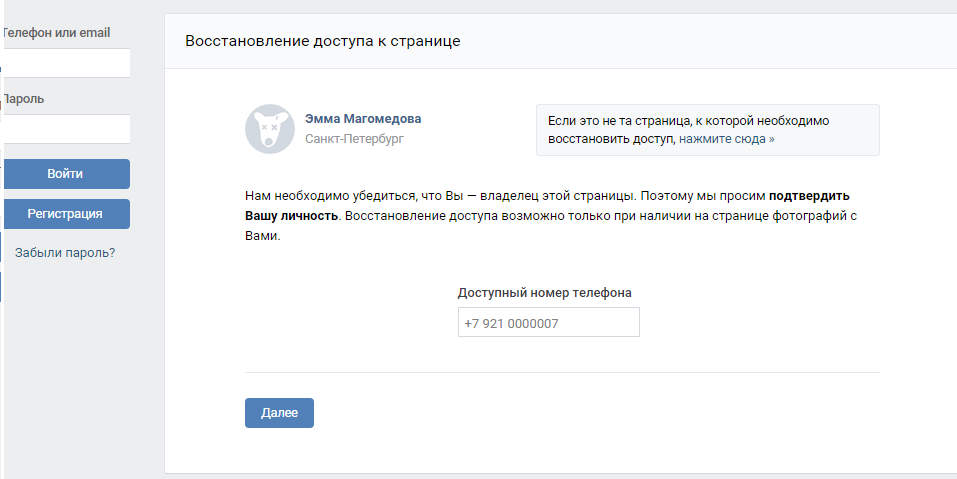 et al. Большие делеции, вызванные расщеплением Cas9. Природа 560 , E8–E9 (2018).
et al. Большие делеции, вызванные расщеплением Cas9. Природа 560 , E8–E9 (2018).
Артикул КАС пабмед Google Scholar
Косицки М., Томберг К. и Брэдли А. Репарация двухцепочечных разрывов, индуцированная CRISPR-Cas9, приводит к большим делециям и сложным перестройкам. Нац. Биотехнолог. 36 , 765–771 (2018).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Leibowitz, M.L. et al. Хромотрипсис как целевое следствие редактирования генома CRISPR-Cas9. Нац. Жене. 53 , 895–905 (2021).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Kim, Y.B. et al. Увеличение масштаба нацеливания на геном и точность редактирования базы с помощью сконструированных слияний Cas9-цитидиндезаминазы. Нац. Биотехнолог. 35 , 371–376 (2017).
Нац. Биотехнолог. 35 , 371–376 (2017).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Gaudelli, N.M. et al. Программируемое редактирование оснований A•T в G•C в геномной ДНК без расщепления ДНК. Природа 551 , 464–471 (2017).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Anzalone, A.V. et al. Редактирование генома с помощью поиска и замены без двухцепочечных разрывов или донорской ДНК. Природа 576 , 149–157 (2019).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Anzalone, A.V. et al. Программируемое удаление, замена, интеграция и инверсия больших последовательностей ДНК с двойным простым редактированием. Нац. Биотехнолог. 40 , 731–740 (2021).
40 , 731–740 (2021).
Артикул пабмед ПабМед Центральный Google Scholar
Yarnall, M.T.N. et al. Вставка больших последовательностей в геном методом перетаскивания без расщепления двухцепочечной ДНК с использованием интеграз, управляемых CRISPR. Нац. Биотехнолог. https://doi.org/10.1038/s41587-022-01527-4 (2022).
Артикул пабмед Google Scholar
Нальдини, Л., Троно, Д. и Верма, И. М. Лентивирусные векторы, два десятилетия спустя. Наука 353 , 1101–1102 (2016).
Артикул КАС пабмед Google Scholar
Querques, I. et al. Высокорастворимая транспозаза «Спящая красавица» улучшает контроль вставки генов. Нац. Биотехнолог. 37 , 1502–1512 (2019).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Юса, К., Чжоу, Л., Ли, М.А., Брэдли, А. и Крейг, Н.Л. Гиперактивная транспозаза piggyBac для применения на млекопитающих. Проц. Натл акад. науч. США 108 , 1531–1536 (2011).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Типани Дж., Вандендрише Т. и Чуа М.К. Транспозоны: переход от доклинических исследований к клиническим испытаниям. Гул. Джин Тер. 28 , 1087–1104 (2017).
Артикул КАС пабмед Google Scholar
Гайдуков Л. и др. Платформа интеграции ДНК с несколькими посадочными площадками для инженерии клеток млекопитающих. Рез. нуклеиновых кислот. 46 , 4072–4086 (2018).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Durrant, M. G. et al. Систематическое открытие рекомбиназ для эффективной интеграции больших последовательностей ДНК в геном человека. Нац. Биотехнолог. https://doi.org/10.1038/s41587-022-01494-w (2022).
G. et al. Систематическое открытие рекомбиназ для эффективной интеграции больших последовательностей ДНК в геном человека. Нац. Биотехнолог. https://doi.org/10.1038/s41587-022-01494-w (2022).
Артикул пабмед Google Scholar
Хью, Б. Э., Сато, Р., Мауро, Д., Стойчев, И. и Оуэнс, Дж. Б. РНК-управляемая транспозиция piggyBac в клетках человека. Синтез. биол. 4 , ysz018 (2019).
Артикул КАС Google Scholar
Ковач, А. и др. РНК-управляемое перенацеливание транспозиции Sleeping Beauty в клетки человека. eLife 9 , e53868 (2020).
Артикул пабмед ПабМед Центральный Google Scholar
Луо В. и др. Сравнительный анализ химерных транспозаз ZFP-, TALE- и Cas9- piggyBac на интеграцию в единый локус в клетках человека. Рез. нуклеиновых кислот. 45 , 8411–8422 (2017).
Рез. нуклеиновых кислот. 45 , 8411–8422 (2017).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Чен, С. П. и Ван, Х. Х. Разработанная система Cas-Transposon для программируемых и сайт-направленных транспозиций ДНК. CRISPR J. 2 , 376–394 (2019).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Klompe, S.E., Vo, PLH, Halpin-Healy, T.S. & Sternberg, S.H. Системы CRISPR-Cas, кодируемые транспозонами, обеспечивают прямую интеграцию ДНК под управлением РНК. Природа 571 , 219–225 (2019).
Артикул КАС пабмед Google Scholar
Vo, P.L.H. et al. CRISPR РНК-управляемые интегразы для высокоэффективной мультиплексной инженерии бактериального генома. Нац. Биотехнолог. 39 , 480–489 (2021).
Нац. Биотехнолог. 39 , 480–489 (2021).
Артикул КАС пабмед Google Scholar
Klompe, S.E. et al. Эволюционное и механистическое разнообразие CRISPR-ассоциированных транспозонов типа I-F. Мол. Ячейка 82 , 616–628 (2022).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Кэмерон, П. и др. Использование систем CRISPR-Cas типа I для инженерии генома в клетках человека. Нац. Биотехнолог. 37 , 1471–1477 (2019).
Артикул КАС пабмед Google Scholar
Chen, Y. et al. Перепрофилирование системы CRISPR-Cas типа I-F в качестве инструмента активации транскрипции в клетках человека. Нац. коммун. 11 , 3136 (2020).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Пикар-Оливер, А. и др. Направленная модуляция транскрипции с помощью систем CRISPR-Cas I типа в клетках человека. Нац. Биотехнолог. 37 , 1493–1501 (2019).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Долан, А. Э. и др. Представление спектра дальних геномных делеций в эмбриональных стволовых клетках человека с использованием CRISPR-Cas типа I. Мол. Сотовый 74 , 936–950 (2019).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Young, J.K. et al. Перепрофилирование CRISPR-каскада типа I-E для активации генов в растениях. Комм. биол. 2 , 383 (2019).
Артикул пабмед ПабМед Центральный Google Scholar
Strecker, J. et al. Вставка ДНК под управлением РНК с CRISPR-ассоциированными транспозазами. Наука 364 , 48–53 (2019).
Наука 364 , 48–53 (2019).
Артикул Google Scholar
Сайто, М. и др. Двойной режим CRISPR-ассоциированного самонаведения транспозонов. Cell 184 , 2441–2453.e18 (2021).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Vo, P.L.H., Acree, C., Smith, M.L. & Sternberg, S.H. Беспристрастное профилирование продуктов транспозиции, управляемой CRISPR РНК, путем секвенирования с длинным считыванием. Моб. ДНК 12 , 13 (2021).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Халпин-Хили, Т. С., Кломпе, С. Э., Штернберг, С. Х. и Фернандес, И. С. Структурная основа нацеливания ДНК с помощью системы CRISPR-Cas, кодируемой транспозоном. Природа 577 , 271–274 (2020).
Артикул КАС пабмед Google Scholar
Питерс, Дж. Э. Tn7. Микробиолог. Спектр. https://doi.org/10.1128/microbiolspec.MDNA3-0010-2014 (2014).
Hoffmann, F. T. et al. Селективное рекрутирование TnsC повышает точность РНК-управляемой транспозиции. Природа 609 , 384–393 (2022).
Артикул КАС пабмед Google Scholar
Behler, J. & Hess, W. R. Подходы к изучению биогенеза РНК CRISPR и ключевых участников. Методы 172 , 12–26 (2020).
Артикул КАС пабмед Google Scholar
Щелкун, М. Д., Тихомирова, М. С., Синкунас, Т., Гасюнас, Г. и Карвелис, Т. Прямое наблюдение за образованием R-петли с помощью одиночных РНК-управляемых эффекторных комплексов Cas9 и Cascade. Проц. Натл акад. науч. США 111 , 9798–9803 (2014 г.).
Проц. Натл акад. науч. США 111 , 9798–9803 (2014 г.).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Gilbert, L. A. et al. CRISPR-опосредованная модульная РНК-управляемая регуляция транскрипции у эукариот. Cell 154 , 442–451 (2013).
Артикул пабмед ПабМед Центральный Google Scholar
Мали, П. и др. Активаторы транскрипции CAS9 для скрининга специфичности мишени и парные никазы для совместной инженерии генома. Нац. Биотехнолог. 31 , 833–838 (2013).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Perez-pinera, P. et al. Активация генов под управлением РНК факторами транскрипции на основе CRISPR-Cas9. Нац. Методы 10 , 973–976 (2013).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Таненбаум, М. Э., Гилберт, Л. А., Ци, Л. С., Вайсман, Дж. С. и Вейл, Р. Д. Система мечения белков для усиления сигнала при экспрессии генов и флуоресцентной визуализации. Cell 159 , 635–646 (2014).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Konermann, S. et al. Активация транскрипции в масштабе генома с помощью сконструированного комплекса CRISPR-Cas9. Природа 517 , 583–588 (2015).
Артикул КАС пабмед Google Scholar
Park, J. U. et al. Структурная основа для выбора сайта-мишени в системах транспозиции ДНК, управляемых РНК. Наука 373 , 768–774 (2021).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Querques, I., Schmitz, M., Oberli, S., Chanez, C. & Jinek, M. Выбор и ремоделирование целевого сайта с помощью систем CRISPR-транспозонов типа V. Природа 599 , 49–502 (2021).
Артикул Google Scholar
Чавес, А. и др. Высокоэффективное Cas9-опосредованное программирование транскрипции. Нац. Методы 12 , 326–3228 (2015).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Thakore, P. I. et al. Высокоспецифичное редактирование эпигенома репрессорами CRISPR-Cas9 для подавления дистальных регуляторных элементов. Нац. Методы 12 , 1143–1149 (2015).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Ван, Т., Ларчер, Л. М., Ма, Л. и Виду, Р. Н. Систематический скрининг широко используемых коммерческих реагентов для трансфекции на предмет эффективной трансфекции одноцепочечных олигонуклеотидов. Молекулы 23 , 2564 (2018).
Молекулы 23 , 2564 (2018).
Артикул пабмед ПабМед Центральный Google Scholar
Walker, M.W.G., Klompe, S.E., Zhang, D.J. & Sternberg, S.H. Библиотеки мутагенеза транспозонов раскрывают новые молекулярные требования во время интеграции ДНК с помощью CRISPR РНК. Препринт на https://www.biorxiv.org/content/10.1101/2023.01.19.524723v1 (2023).
Сарновский, Р. Дж., Мэй, Э. У. и Крейг, Н. Л. Транспозаза Tn7 представляет собой гетеромерный комплекс, в котором активность разрыва и соединения ДНК распределена между различными генными продуктами. EMBO J. 15 , 6348–6361 (1996).
Артикул КАС пабмед ПабМед Центральный Google Scholar
North, S. H. & Nakai, H. Факторы хозяина, которые способствуют разборке транспозосом и пути PriA-PriC для повторной сборки примосом. Мол. микробиол. 56 , 1601–1616 (2005).
Мол. микробиол. 56 , 1601–1616 (2005).
Артикул КАС пабмед Google Scholar
Adeyemi, R. O. et al. Комплекс Protexin противодействует резекции застопорившихся вилок, способствуя гомологичной рекомбинации и репарации поперечных связей. Мол. Сотовый 81 , 4440–4456 (2021 г.).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Чичча, А. и Элледж, С. Дж. Реакция на повреждение ДНК: сделать игру с ножами безопасной. Мол. Ячейка 40 , 179–204 (2010).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Холдер, Дж. В. и Крейг, Н. Л. Архитектура посттранспозиционного комплекса Tn7: сложная нуклеопротеиновая структура. Дж. Мол. биол. 401 , 167–181 (2010).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Левченко И., Луо Л. и Бейкер Т. А. Разборка тетрамера Mu-транспозазы шапероном ClpX. Гены Дев. 9 , 2399–2408 (1995).
Артикул КАС пабмед Google Scholar
Круклитис Р., Велти Д. Дж. и Накаи Х. Белок ClpX из Escherichia coli активирует транспозазу Mu бактериофага в комплексе переноса цепи для инициации синтеза ДНК Mu. EMBO J. 15 , 935–944 (1996).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Мхаммеди-Алаул, А., Пато, М. и Гама, М.-Дж. и Туссен, А. Новый компонент механизма репликативной транспозиции бактериофага Mu: Escherichia coli Белок ClpX. Мол. микробиол. 11 , 1109–1116 (1994).
Артикул Google Scholar
Abdelhakim, A.H., Oakes, E.C., Sauer, R.T. & Baker, T.A. Уникальные контакты направляют высокоприоритетное распознавание комплекса тетрамерной Mu-транспозазы-ДНК с помощью AAA+-унфолдазы ClpX. Мол. Cell 30 , 39–50 (2008).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Sauer, R. T. & Baker, T. A. ААА+ протеазы: работающие на АТФ машины разрушения белков. год. Преподобный Биохим. 80 , 587–612 (2011).
Артикул КАС пабмед Google Scholar
Левченко И., Ямаути М. и Бейкер Т. А. ClpX и MuB взаимодействуют с перекрывающимися областями транспозазы Mu: последствия для контроля пути транспозиции. Гены Дев. 11 , 1561–1572 (1997).
Артикул КАС пабмед Google Scholar
Baker, T. A. & Sauer, R. T. ClpXP, машина для разворачивания и расщепления белка, работающая на АТФ. Биохим. Биофиз. Акта Мол. Сотовый рез. 1823 , 15–28 (2012).
Артикул КАС Google Scholar
Херш, Г. Л., Бертон, Р. Э., Болон, Д. Н., Бейкер, Т. А. и Зауэр, Р. Т. Асимметричные взаимодействия АТФ с разворачиванием AAA+ ClpX6: аллостерический контроль белковой машины. Cell 121 , 1017–1027 (2005).
Артикул КАС пабмед Google Scholar
Джоши С.А., Херш Г.Л., Бейкер Т.А. и Зауэр Р.Т. Связь между ClpX и ClpP во время обработки и деградации субстрата. Нац. Структура Мол. биол. 11 , 404–411 (2004).
Артикул КАС пабмед Google Scholar
Сиддики С. М., Зауэр Р. Т. и Бейкер Т. А. Роль процессирующей поры ClpX AAA+ АТФазы в распознавании и задействовании специфических белковых субстратов. Гены Дев. 18 , 369–374 (2004).
А. Роль процессирующей поры ClpX AAA+ АТФазы в распознавании и задействовании специфических белковых субстратов. Гены Дев. 18 , 369–374 (2004).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Strecker, J., Zhang, F. & Ladha, A. Системы CRISPR-ассоциированных транспозаз и способы их использования. https://patents.google.com/patent/WO2020131862A1/en (2020 г.).
Tou, C. J., Orr, B. & Kleinstiver, B. P. Точная вставка ДНК путем вырезания и вставки с использованием сконструированных CRISPR-ассоциированных транспозаз типа V-K. Нац. Биотехнолог. https://doi.org/10.1038/s41587-022-01574-x (2023).
Артикул пабмед Google Scholar
Озджан, А. и др. Программируемое нацеливание РНК с однобелковым эффектором CRISPR Cas7-11. Природа 597 , 720–725 (2021).
Артикул пабмед Google Scholar
Shen, Y. et al. Структурная основа нацеливания ДНК транспозоном Tn7. Нац. Структура Мол. биол. 29 , 143–151 (2022).
Артикул КАС пабмед Google Scholar
Гу, Б. и др. Связанные с транскрипцией изменения ядерной подвижности цис-регуляторных элементов млекопитающих. Наука 359 , 1050–1055 (2018).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Чен Б., Гуан Дж. и Хуанг Б. Визуализация специфической геномной ДНК в живых клетках. год. Преподобный Биофиз. 45 , 1–23 (2016).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Schmitz, M. , Querques, I., Oberli, S., Chanez, C. & Jinek, M. Структурная основа для сборки CRISPR-ассоциированного комплекса транспозонов типа V. Сотовый 185 , 4999–5010 (2022).
, Querques, I., Oberli, S., Chanez, C. & Jinek, M. Структурная основа для сборки CRISPR-ассоциированного комплекса транспозонов типа V. Сотовый 185 , 4999–5010 (2022).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Фрике, Т. и др. Целевой нокдаун РНК комплексом CRISPR-Cas типа III у рыбок данио. CRISPR J. 3 , 299–313 (2020).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Petassi, M.T., Hsieh, S. & Peters, J.E. Категоризация направляющей РНК позволяет выбирать сайт-мишень в транспозонах Tn7-CRISPR-Cas. Cell 183 , 1757–1771 (2020).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Yeo, N.C. et al. Усовершенствованный репрессор CRISPR для целенаправленной регуляции генов млекопитающих. Нац. Методы 15 , 611–616 (2018).
Нац. Методы 15 , 611–616 (2018).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Lee, T.I., Johnstone, S.E. & Young, R.A. Иммунопреципитация хроматина и анализ местоположения белка на основе микрочипов. Нац. протокол 1 , 729–748 (2006).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Weinberg, D. N. et al. Метка гистонов h4K36me2 рекрутирует DNMT3A и формирует ландшафт межгенного метилирования ДНК. Природа 573 , 281–286 (2019).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Цю, X. и др. CoBRA: Контейнерный рабочий процесс биоинформатики для воспроизводимого анализа ChIP/ATAC-seq. Геномика Протеомика Биоинформатика 19 , 652–661 (2021).
Артикул пабмед ПабМед Центральный Google Scholar
Ли, Х. и др. Формат Sequence Alignment/Map и SAMtools. Биоинформатика 25 , 2078–2079 (2009).
Артикул пабмед ПабМед Центральный Google Scholar
Zhang, Y. et al. Модельный анализ ChIP-Seq (MACS). Геном Биол. 9 , R137 (2008).
Артикул пабмед ПабМед Центральный Google Scholar
Робинсон, Дж. Т. и др. Интегративный просмотрщик геномики. Нац. Биотехнолог. 29 , 24–26 (2011).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Куинлан, А. Р. и Холл, И. М. BEDTools: гибкий набор утилит для сравнения геномных признаков.

 Это необходимо для того, чтобы администрация смогла вас идентифицировать и вернуть доступ к аккаунту.
Это необходимо для того, чтобы администрация смогла вас идентифицировать и вернуть доступ к аккаунту.