Дыхательная недостаточность — Пульмонология | Медицинский центр «САДКО» Нижний Новгород
Дыхательная недостаточность (ДН) – ограничение способности легких обеспечивать нормальный газовый состав артериальной крови, происходит перенапряжение компенсаторных возможностей системы внешнего дыхания.
Причины возникновения:
- острые и хронические заболевания бронхо-легочной системы
- поражения ЦНС
- малокровие (анемия)
- гипертензия в малом круге кровообращения,
- сосудистая патология легких и сердца,
- опухоли легких
Симптомы и виды
Классическими признаками дыхательной недостаточности служат:
- синдром слабости и утомления дыхательной мускулатуры;
- одышка.
По темпам наступления патологического состояния выделяют острую и хроническую ДН.
Острая дыхательная недостаточность развивается в течение короткого времени, в течение нескольких минут или часов и требует неотложных терапевтических мероприятий.
Симптоматика:
- нарастающая одышка.
- Нарушение центральной регуляции дыхания, наблюдаются участие в дыхании лишь мышц шеи и движение гортани
- чувство беспокойства и возбуждения, неадекватное поведение.
- Заторможенность, постепенная потеря сознания
- судороги,
- землистый оттенок кожи
На начальных стадиях отмечается тахикардия, тенденция к повышению артериального давления.
Хроническая дыхательная недостаточность чаще всего развивается при хронических обструктивных заболеваниях легких (хронический обструктивный бронхит), ожирении, резекции легких, кифосколиозе. При всех перечисленных ситуациях возникающая гипоксия приводит к увеличению работы дыхательной мускулатуры, что в течение некоторого времени обеспечивает сохранение газового состава крови.
Симптоматика:
- постепенно развивающаяся одышка.
- одышку уже при незначительных усилиях или даже в покое
- сонливость
- отмечается расширение сосудов кожи лица.

- Конечности с багрово-синюшным оттенком
- появляется одутловатость лица.
Основные причины возникновения дыхательной недостаточности — острые и хронические заболевания лёгких, ведущие к гиповентиляции обструктивного типа.
Осложнения дыхательной недостаточности
Дыхательная недостаточность — неотложное, угрожающее здоровью и жизни человека состояние. При неоказании своевременной помощи острая дыхательная недостаточность может привести к гибели пациента.
Длительное течение и прогрессирование хронической дыхательной недостаточности приводит к развитию правожелудочковой сердечной недостаточности в результате дефицита снабжения сердечной мышцы кислородом и ее постоянных перегрузок.
Диагностика
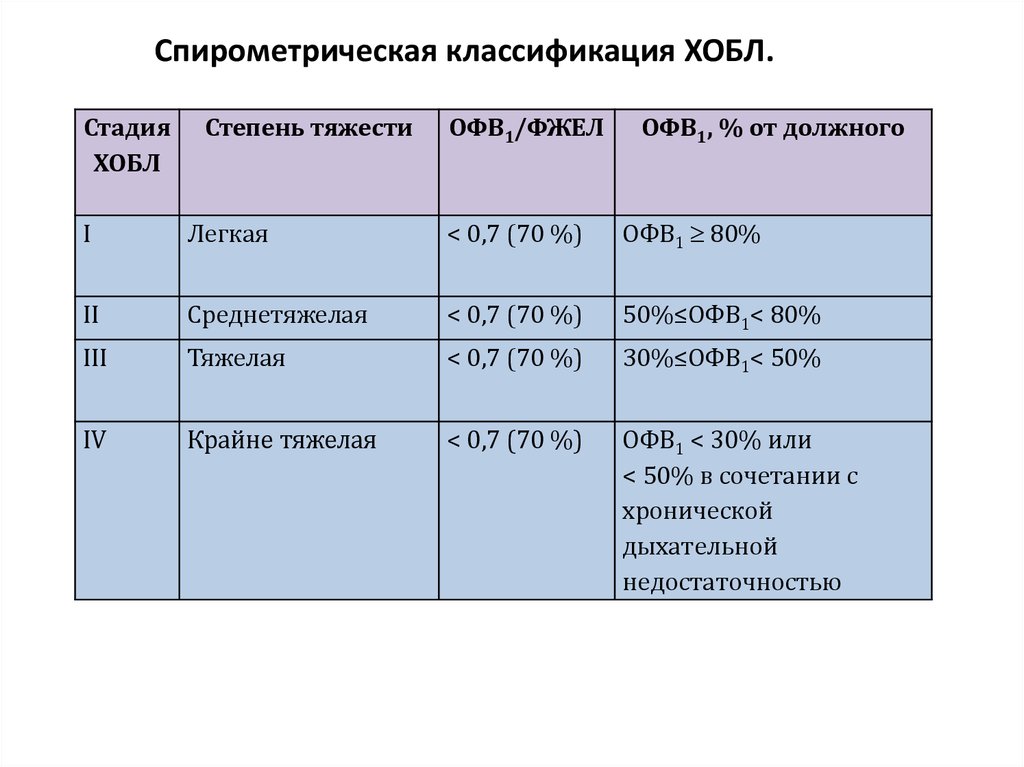
- Функциональная диагностика внешнего дыхания, позволяющая провести оценку вентиляционной способности легких.
 При этом измеряется жизненная емкость легких, минутный объем дыхания, скорость движения воздуха по различным отделам дыхательных путей.
При этом измеряется жизненная емкость легких, минутный объем дыхания, скорость движения воздуха по различным отделам дыхательных путей. - Лабораторный анализ газового состава крови, позволяющий определить степень насыщения артериальной крови кислородом и углекислым газом
- Рентгенография органов грудной клетки с целью выявления поражения грудной клетки, легких, сосудов, бронхов.
Лечение и профилактика
При лечении больных острой ДН следует проводить неотложную терапию (терапию жизнеобеспечения), включающую обеспечение и поддерживание проходимости дыхательных путей, кислородотерапию, вспомогательную вентиляцию или ИВЛ. При болях, ограничивающих экскурсии грудной клетки, назначают анальгетики , новокаиновую блокаду.
Лечение хронической ДН является симптоматическим и сводится главным образом к контролю прогрессирования основного заболевания, своевременному купированию обострений хронического обструктивного бронхита (наиболее частая причина хронической ДН). Особенно эффективна кислородотерапия. Обязателен отказ от курения. Всем больным показана лечебная физкультура и дыхательная гимнастика со специальным комплексом дыхательных упражнений.
Особенно эффективна кислородотерапия. Обязателен отказ от курения. Всем больным показана лечебная физкультура и дыхательная гимнастика со специальным комплексом дыхательных упражнений.
Страница не найдена
Размер:
AAAЦвет: C C C
Изображения Вкл. Выкл.
Обычная версия сайта
RUENBY
Гомельский государственный
медицинский университет
- Университет
- Университет
- История
- Руководство
- Устав и Символика
- Воспитательная деятельность
- Организация образовательного процесса
- Международное сотрудничество
- Система менеджмента качества
- Советы
- Факультеты
- Кафедры
- Подразделения
- Первичная профсоюзная организация работников
- Издания университета
- Гордость университета
- Выпускник-2021
- Первичная организация «Белорусский союз женщин»
- Одно окно
- ГомГМУ в международных рейтингах
- Структура университета
- Абитуриентам
- Приёмная комиссия
- Университетская олимпиада по биологии
- Целевая подготовка
- Заключение, расторжение «целевого» договора
- Льготы для молодых специалистов
- Архив проходных баллов
- Карта и маршрут проезда
- Порядок приёма на 2023 год
- Специальности
- Контрольные цифры приёма в 2022 году
- Стоимость обучения
- Информация о ходе приёма документов
- Приём документов и время работы приёмной комиссии
- Порядок приёма граждан РФ, Кыргызстана, Таджикистана, Казахстана
- Горячая линия по вопросам вступительной кампании
- Студентам
- Первокурснику
- Расписание занятий
- Расписание экзаменов
- Информация для студентов
- Студенческий клуб
- Спортивный клуб
- Общежитие
- Нормативные документы
- Практика
- Стоимость обучения
- Безопасность жизнедеятельности
- БРСМ
- Профком студентов
- Учебный центр практической подготовки и симуляционного обучения
- Многофункциональная карточка студента
- Анкетирование студентов
- Выпускникам
- Интернатура и клиническая ординатура
- Докторантура
- Аспирантура
- Магистратура
- Распределение
- Врачам и специалистам
- Профессорский консультативный центр
- Факультет повышения квалификации и переподготовки
- Иностранным гражданам
- Факультет иностранных студентов
- Стоимость обучения
- Регистрация и визы
- Полезная информация
- Правила приёма
- Информация о возможностях и условиях приема в 2022 году
- Официальные представители ГомГМУ по набору студентов
- Страхование иностранных граждан
- Приём на Подготовительное отделение иностранных граждан
- Прием иностранных граждан для обучения на английском языке / Training of foreign students in English
- Повышение квалификации и переподготовка для иностранных граждан
- Научная деятельность
- Направления научной деятельности
- Научно-педагогические школы
- Инновационные технологии в ГомГМУ
- Научно-исследовательская часть
- Научно-исследовательская лаборатория
- Конкурсы, гранты, стипендии
- Научные мероприятия
- Работа комитета по этике
- В помощь исследователю
- Совет молодых ученых
- Студенчеcкое научное общество
- Диссертационный совет
- Патенты
- Инструкции на метод
- «Горизонт Европа»
- Госпрограмма (ЧАЭС)
- Главная
One-Zero (2009) — IMDb
- Cast & crew
- User reviews
IMDbPro
Original title: Wahed-Sefr
- 20092009
- 1h 45m
IMDb RATING
6. 4/10
4/10
544
ВАША ОЦЕНКА
8 персонажей за один день, один город, один футбольный матч, один гол, Египет побеждает один-ноль, все счастливы.. это все настоящее счастье? или это просто выброс подавленных взрывных эмоций всех… Прочитать всех 8 персонажей за один день, один город, один футбольный матч, один гол, Египет побеждает один-ноль, все счастливы.. это все настоящее счастье? или это просто выброс подавленных взрывных эмоций всех 8 персонажей на грани нервного срыва!.8 персонажей за один день, один город, один футбольный матч, один гол, Египет побеждает раз-ноль, все счастливы.. это это все настоящее счастье? или это просто выброс подавленных взрывных эмоций всех 8 персонажей на грани нервного срыва!.
IMDb RATING
6.4/10
544
YOUR RATING
- Kamlah Abu-Zikri
- Mariam Naoum(screenplay)
- Stars
- Ilham Shaheen
- Kal Naga
- Nilli Karim
- Камла Абу-Зикри
- Мариам Наум (Сценарий)
- Звезды
- ilham Shaheen
- Звезды
- ilham Shaheen
- Kal Naga
- Nilli Karim
- Awards
- 5 wins & 3 nominations
Photos
Top cast
Ильхам Шахин
Кал Нага
- Шериф
- (как Халед Абол Нага)
Нилли Карим
- Рихам
- Hassan
- (as Mohamed Alaa Jamaica)
- Kamlah Abu-Zikri
- Mariam Naoum (Сценарий)
- All Cast & Crew
- Производство, Box Office & больше в IMDBPRO
- .
 0004
0004 - MIGOS_IMAGE
- 26 апреля 2009
1 час 45 минут
- 1.
 37 : 1
37 : 1
- 1.
- Программное обеспечение
- Открытый доступ
- Опубликовано:
- Ф. Александр Вольф ORCID: orcid.org/0000-0002-8760-7838 1 ,
- Филипп Ангерер 1 и
- Фабиан Дж. Тайс 1,2
70 тыс.
 обращений
обращений1427 цитирований
199 Альтметрический
Сведения о показателях
Анализ 68 579 РВМС (рис. 1) в сравнении с набором Cell Ranger R [6]: https://github.com/theislab/scanpy_usage/tree/master/170503_zheng17.
Кластеризация и идентификация типов клеток, адаптировано и проверено на основе http://satijalab.org/seurat/pbmc3k_tutorial.htmlи одного из учебных пособий Seurat [2]: https://github.com/theislab/scanpy_usage/tree/master/170505_seurat .
Визуализация и кластеризация 1,3 миллиона ячеек (рис.
 1c): https://github.com/theislab/scanpy_usage/tree/master/170522_visualizing_one_million_cells.
1c): https://github.com/theislab/scanpy_usage/tree/master/170522_visualizing_one_million_cells.Реконструкция ветвящихся процессов с помощью псевдовремени диффузии [21]: https://github.com/theislab/scanpy_usage/tree/master/170502_haghverdi16.
Моделирование отдельных клеток с использованием сетей регуляции генов [24]: https://github.com/theislab/scanpy_usage/tree/master/170430_krumsiek11.
Анализ результатов глубокого обучения для изображений с одной ячейкой [25]: https://github.com/theislab/scanpy_usage/tree/master/170529_images.
Сатия Р., Фаррелл Дж. А., Геннерт Д., Шир А. Ф., Регев А. Пространственная реконструкция данных экспрессии генов одноклеточных. Нац биотехнолог. 2015 г.; 33:495–502.
Артикул КАС пабмед ПабМед Центральный Google Scholar
Trapnell C, et al. Динамика и регуляторы решений клеточных судеб выявляются с помощью псевдовременного упорядочения одиночных клеток. Нац биотехнолог. 2014; 32:381–6.
Артикул КАС пабмед ПабМед Центральный Google Scholar
Харченко П.В., Зильберштейн Л., Скадден Д.Т. Байесовский подход к анализу дифференциальной экспрессии одноклеточных. Нат Методы. 2014; 11: 740–2.
Артикул КАС пабмед ПабМед Центральный Google Scholar
Finak, G, et al.
 MAST: гибкая статистическая основа для оценки транскрипционных изменений и характеристики гетерогенности в данных секвенирования одноклеточной РНК. Геном биол. 2015 г.; 16:278.
MAST: гибкая статистическая основа для оценки транскрипционных изменений и характеристики гетерогенности в данных секвенирования одноклеточной РНК. Геном биол. 2015 г.; 16:278.Артикул Google Scholar
Zheng GXY и др. Массивно параллельное цифровое профилирование транскрипции отдельных клеток. Нац коммун. 2017; 8:14049.
Артикул КАС пабмед ПабМед Центральный Google Scholar
Маккарти Д., Уиллс К., Кэмпбелл К. Скейтер: набор инструментов для анализа отдельных клеток для данных об экспрессии генов в R. Bioinformatics. 2017; 33:1179.
ПабМед ПабМед Центральный Google Scholar
Лун А., Маккарти Д., Мариони Дж. Пошаговый рабочий процесс низкоуровневого анализа данных секвенирования РНК одиночных клеток с помощью Bioconductor.
 F1000Исследование. 2016; 5:2122.
F1000Исследование. 2016; 5:2122.ПабМед ПабМед Центральный Google Scholar
Абади М. и др. TensorFlow: крупномасштабное машинное обучение в гетерогенных системах. 2015. https://www.tensorflow.org/about/bib.
Macosko EZ, et al. Высокопараллельное профилирование полногеномной экспрессии отдельных клеток с использованием нанолитровых капель. Клетка. 2015 г.; 161:1202–14.
Артикул КАС пабмед ПабМед Центральный Google Scholar
Coifman RR, et al. Геометрические диффузии как инструмент гармонического анализа и определения структуры данных: карты диффузии. Proc Natl Acad Sci. 2005 г.; 102: 7426–31.
Артикул КАС пабмед ПабМед Центральный Google Scholar
Amir EAD, Davis KL, Tadmor MD, Simonds EF, Levine JH, Bendall SC, et al.
 ViSNE позволяет визуализировать высокоразмерные данные по отдельным клеткам и выявляет фенотипическую гетерогенность лейкемии. Нац биотехнолог. 2013; 31: 545–52.
ViSNE позволяет визуализировать высокоразмерные данные по отдельным клеткам и выявляет фенотипическую гетерогенность лейкемии. Нац биотехнолог. 2013; 31: 545–52.Артикул КАС ПабМед Центральный Google Scholar
Рейнгольд Э.М. Рисование графика с помощью силового размещения. Программное обеспечение Практика Exp. 1991 год; 21:1129–64.
Артикул Google Scholar
Чарди Г., Непуш Т. Программный пакет igraph для сложных сетевых исследований. InterJournal Compl Syst. 2006 г.; 2006:1695.
Google Scholar
Weinreb C, Wolock S, Klein A. Spring: кинетический интерфейс для визуализации многомерных данных экспрессии отдельных клеток. bioRxiv. 2017. https://doi.org/10.1093/bioinformatics/btx792.
Бюттнер Ф., Тайс Ф.
 Дж. Карты диффузии для многомерного одноклеточного анализа данных дифференцировки. Биоинформатика. 2015 г.; 31:2989–98.
Дж. Карты диффузии для многомерного одноклеточного анализа данных дифференцировки. Биоинформатика. 2015 г.; 31:2989–98.Артикул пабмед Google Scholar
Angerer P, et al.destiny: Карты диффузии для крупномасштабных одноклеточных данных в R. Bioinformatics. 2015 г.; 32:1241.
Артикул пабмед Google Scholar
Блондель В.Д., Гийом Ж-Л., Ламбиот Р., Лефевр Э. Быстрое развертывание сообществ в больших сетях. J Стат Мех. 2008 г.; 2008: P10008.
Артикул Google Scholar
Levine JH, et al. Фенотипическое вскрытие ОМЛ на основе данных выявляет клетки, подобные предшественникам, которые коррелируют с прогнозом. Клетка. 2015 г.; 162:184–97.
Артикул пабмед ПабМед Центральный Google Scholar
Xu C, Su Z.
 Идентификация типов клеток по одноклеточным транскриптомам с использованием нового метода кластеризации. Биоинформатика. 2015 г.; 31:1974–80.
Идентификация типов клеток по одноклеточным транскриптомам с использованием нового метода кластеризации. Биоинформатика. 2015 г.; 31:1974–80.Артикул КАС пабмед Google Scholar
Хагверди Л., Бюттнер М., Вольф Ф.А., Бюттнер Ф., Тайс Ф.Дж. Диффузионное псевдовремя надежно реконструирует ветвящиеся клеточные линии. Нат Методы. 2016; 13:845–8.
Артикул пабмед Google Scholar
Qiu X, et al. Встраивание обратного графа разрешает сложные одноклеточные траектории. Нат Методы. 2017; 14:979–82.
Артикул КАС пабмед Google Scholar
Setty, M, et al. Wishbone идентифицирует бифуркационные траектории развития по данным одноклеточных. Нац биотехнолог. 2016; 34: 637–45.
Артикул Google Scholar
Wittmann, DM, et al.
 Преобразование булевых моделей в непрерывные модели: методология и применение к передаче сигналов Т-клеточного рецептора. BMC Сист Биол. 2009 г.; 3:98.
Преобразование булевых моделей в непрерывные модели: методология и применение к передаче сигналов Т-клеточного рецептора. BMC Сист Биол. 2009 г.; 3:98.Артикул Google Scholar
Эйленберг П. и др. Реконструкция клеточного цикла и прогрессирования заболевания с помощью глубокого обучения. Нац коммун. 2017; 8:463.
Артикул пабмед ПабМед Центральный Google Scholar
Huber, W, et al. Организация высокопроизводительного геномного анализа с помощью Bioconductor. Нат Методы. 2015 г.; 12:115–21.
Артикул Google Scholar
Педрегоса Ф. и др. Scikit-learn: машинное обучение в Python. Дж. Мах Узнать Рез. 2011 г.; 12:2825–30.
Google Scholar
Хагберг А.А., Шульт Д.
 А., Сварт П.Дж. Изучение структуры, динамики и функций сети с помощью networkx. В: Материалы 7-й конференции Python в науке (SciPy2008). Пасадена: 2008. с. 11–15.
А., Сварт П.Дж. Изучение структуры, динамики и функций сети с помощью networkx. В: Материалы 7-й конференции Python в науке (SciPy2008). Пасадена: 2008. с. 11–15.Bastian M, Heymann S, Jacomy M. Gephi: программное обеспечение с открытым исходным кодом для исследования сетей и управления ими. Международная конференция AAAI по блогам и социальным сетям. 2009.
Angerer, P, et al. Отдельные клетки создают большие данные: новые проблемы и возможности в транскриптомике. Curr Opin Syst Biol. 2017; 4:85–91.
Артикул Google Scholar
Регев А. и др. Научный форум: атлас клеток человека. электронная жизнь. 2017; 6:e27041.
Артикул пабмед ПабМед Центральный Google Scholar
Lun ATL, Pages̀ H, Smith ML. beachmat: API-интерфейс Bioconductor C++ для доступа к данным геномики отдельных клеток из различных типов матрицы R.
 bioRxiv. 2017. https://doi.org/10.1101/167445.
bioRxiv. 2017. https://doi.org/10.1101/167445.ван дер Уолт С., Колберт С.К., Вароко Г. Массив NumPy: структура для эффективных численных вычислений. Инженер по вычислительной технике. 2011 г.; 13:22–30.
Артикул Google Scholar
Джонс Э., Олифант Т., Петерсон П. и др. SciPy: научные инструменты с открытым исходным кодом для Python. 2001. https://www.scipy.org/citing.html.
Хантер Д.Д. Matplotlib: среда 2D-графики. Инженер по вычислительной технике. 2007 г.; 9:90–5.
Артикул Google Scholar
МакКинни В. Структуры данных для статистических вычислений в Python В: ван дер Уолт С., Миллман Дж., редакторы. Материалы 9-й конференции Python в науке: 2010. с. 51–6.
Коллетт А. Python и HDF5. Sebasto pol: О’Рейли; 2013.

Google Scholar
Сиболд С., Перктольд Дж. Статистические модели: эконометрическое и статистическое моделирование с помощью Python. 9-я Python в научной конференции. 2010.
Waskom, M, et al. В: Varoquaux G, Vaught T, Millman J, (eds).Seaborn; 2016. http://doi.org/10.5281/zenodo.12710, https://networkx.github.io/documentation/networkx-1.10/reference/citing.html.
Ульянов Д. Мультикор-цнэ. 2016. https://github.com/DmitryUlyanov/Multicore-TSNE.
Трааг V, Лувен. Гитхаб. 2017. https://doi.org/10.5281/zenodo.595481.
ЛеКун Ю., Бенжио Ю., Хинтон Г. Глубокое обучение. Природа. 2015 г.; 521: 436–44.
Артикул КАС пабмед Google Scholar
Lippert C, Casale FP, Rakitsch B, Stegle O.
 In: van der Walt S, Millman J, (eds).Limix: генетический анализ множественных признаков; 2014 г. https://doi.org/10.1101/003905, http://conference.scipy.org/proceedings/scipy2010/mckinney.html. bioRxiv.
In: van der Walt S, Millman J, (eds).Limix: генетический анализ множественных признаков; 2014 г. https://doi.org/10.1101/003905, http://conference.scipy.org/proceedings/scipy2010/mckinney.html. bioRxiv.Matthews AGdeG, van der Wilk M, Nickson T, Fujii K, Boukouvalas A, Le’on-Villagr’a P, Ghahramani Z, Hensman J. GPflow: библиотека процессов Гаусса с использованием TensorFlow. Дж. Мах Узнать Рез. 2017; 18(40):1–6. http://jmlr.org/papers/v18/16-537.html.
Мэтьюз де, Г., Александр Г. и др. GPflow: библиотека процессов Гаусса с использованием TensorFlow. Дж. Мах Узнать Рез. 2017; 18:1–6. https://github.com/SheffieldML/GPy.
Buettner F, et al. Вычислительный анализ межклеточной неоднородности в данных секвенирования РНК одной клетки выявляет скрытые субпопуляции клеток. Нац биотехнолог. 2015 г.; 33:155.
Артикул КАС пабмед Google Scholar
Buettner F, Pratanwanich N, McCarthy DJ, Marioni JC, Stegle O.
 f-scLVM: масштабируемый и универсальный факторный анализ для одноклеточной РНК-seq. Геном биол. 2017; 18:212.
f-scLVM: масштабируемый и универсальный факторный анализ для одноклеточной РНК-seq. Геном биол. 2017; 18:212.Артикул пабмед ПабМед Центральный Google Scholar
ДеТомасо Д., Йосеф Н. Fastproject: инструмент для низкоразмерного анализа данных секвенирования РНК одной клетки. БМК Биоинформ. 2016; 17:315.
Артикул Google Scholar
Шекхар К., Бродин П., Дэвис М.М., Чакраборти А.К. Автоматическая классификация клеточной экспрессии с помощью нелинейного стохастического встраивания (акцент): 2013. стр. 202–7.
Dixit A, et al. Perturb-seq: анализ молекулярных цепей с масштабируемым профилированием одноклеточной РНК объединенных генетических скринингов. Клетка. 2016; 167:1853–66.e17.
Артикул КАС пабмед ПабМед Центральный Google Scholar
Свенссон В.
 и др. Анализ мощности экспериментов по секвенированию РНК в одной клетке. Нат Методы. 2017; 14:381.
и др. Анализ мощности экспериментов по секвенированию РНК в одной клетке. Нат Методы. 2017; 14:381.Артикул КАС пабмед ПабМед Центральный Google Scholar
Giecold G, Marco E, Garcia SP, Trippa L, Yuan G-C. Надежная реконструкция родословной на основе многомерных одноклеточных данных. Нуклеиновые Кислоты Res. 2016; 44:e122.
Артикул пабмед ПабМед Центральный Google Scholar
Helmholtz Zentrum München – Немецкий исследовательский центр гигиены окружающей среды, Институт вычислительной биологии, Мюнхен, Нойхерберг, Германия
F. Alexander Wolf, Philipp Angerer & Fabian J. Theis
Департамент математики, Technische Universität München, Munich, Germany
Fabian J. Theis
- Philipp Angerer
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Академия
- Fabian J.
 Theis
TheisПосмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
102 Ahmad El-Fishawi
Intesar
Zeina
Lotfy Labib
Hussien El Imam
Aida Riyad
Mohamed Alaa
Omar El Saeed
Kamel El Sherif
FAHD HASSAN
Подробнее, как это
La Tarago wa la Esteslam
Malek Wa Ketaba
El-Gezirah
Fi Shaket Masr El Gedeeda
Subly Nights
Wannous
Scheherast
Девушка с завода
Ан эль-Ишк ва эль-Хава
Маменькин сынок
Маллаки Искандария
Сюжетная линия
Отзывы пользователей1
Обзор
80 Рекомендуемый обзор9
10/
10
Глубоко и трогательно!
Со времен шедевров Юсефа Шахина не было египетских фильмов с таким совершенством! Обидно! Да, но это правда! В любом случае, 2009 год очень многообещающий с началом «One-Zero».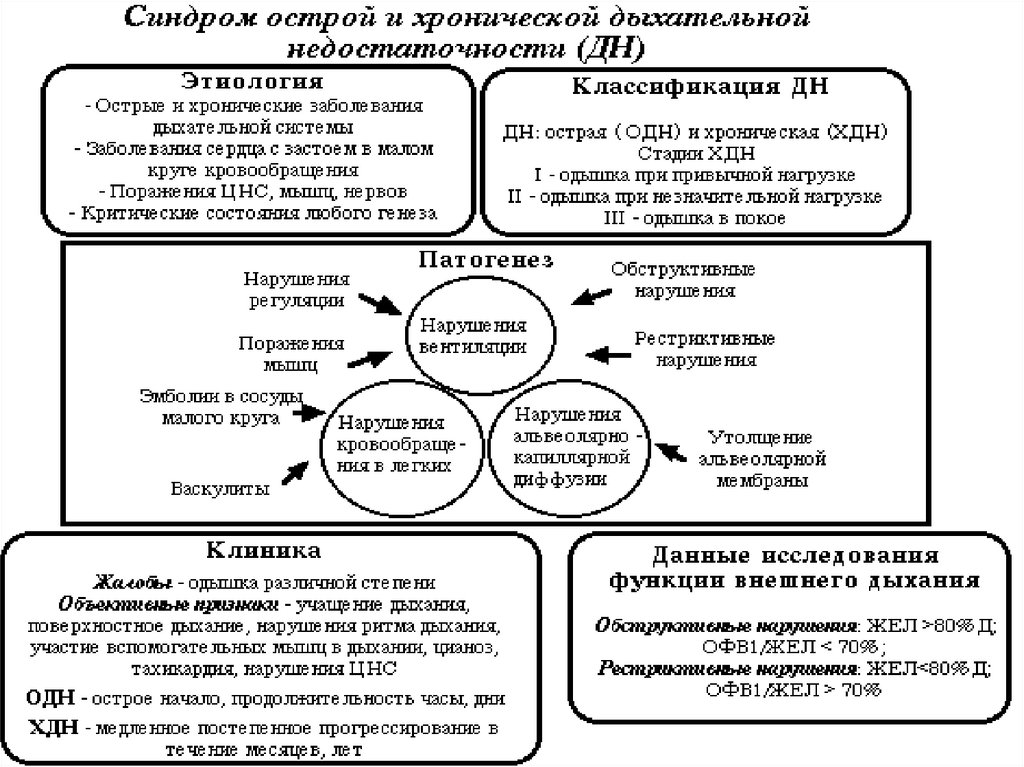 Хорошо сплетенная очень запутанная история о 8 персонажах, живущих в современном Каире, страдающих от его коррупции, религиозных табу, лицемерия и репрессий. 8 с очень разным прошлым в очень напряженный день, а затем связаны со счастьем в конце, когда Победа достигнута. Невероятная история, рассказанная в уникальном стиле, хоть и напоминающая мне шедевр Иньярриту «Вавилон», все же это настоящая египетская история. Два больших пальца вверх!
Хорошо сплетенная очень запутанная история о 8 персонажах, живущих в современном Каире, страдающих от его коррупции, религиозных табу, лицемерия и репрессий. 8 с очень разным прошлым в очень напряженный день, а затем связаны со счастьем в конце, когда Победа достигнута. Невероятная история, рассказанная в уникальном стиле, хоть и напоминающая мне шедевр Иньярриту «Вавилон», все же это настоящая египетская история. Два больших пальца вверх!
Полезно • 7
0
IMDB Best of 2022
IMDB Best of 2022
Откройте для Skyrock Skyrocket на Imdb’s Starteter. Лучшее за 2022 год; включая лучшие трейлеры, плакаты и фотографии.
Подробнее
Подробнее
Кассовые сборы
Технические характеристики
Новости по теме
Добавить страницу
Предложить отредактировать или добавить отсутствующий контент
Ответить
Еще для изучения
Недавно просмотренные
У вас нет недавно просмотренных страниц
SCANPY: крупномасштабный анализ данных экспрессии генов в одной клетке | Геномная биология
7 Биология генома том 19 , номер статьи: 15 (2018) Процитировать эту статью
Abstract
Scanpy — это масштабируемый инструментарий для анализа данных экспрессии генов в одиночных клетках. Он включает в себя методы предварительной обработки, визуализации, кластеризации, вывода псевдовремени и траектории, тестирования дифференциальной экспрессии и моделирования сетей регуляции генов. Его реализация на основе Python эффективно работает с наборами данных из более чем одного миллиона ячеек (https://github.com/theislab/Scanpy). Наряду со Scanpy мы представляем AnnData, универсальный класс для обработки аннотированных матриц данных (https://github. com/theislab/anndata).
com/theislab/anndata).
История вопроса
Простые интегрированные рабочие процессы анализа транскриптомных данных отдельных клеток [1] стали возможными благодаря таким платформам, как SEURAT [2], MONOCLE [3], SCDE/PAGODA [4], MAST [5], CELL RANGER [6], SCATER [7] и SCRAN [8]. Однако эти платформы не масштабируются для все более доступных больших наборов данных, содержащих до миллиона ячеек и более. Здесь мы представляем структуру, которая преодолевает это ограничение и предоставляет аналогичные возможности анализа. Более того, в отличие от существующих фреймворков на основе R, реализация SCANPY на основе Python легко взаимодействует с передовыми пакетами машинного обучения, такими как TENSORFLOW [9].].
Результаты
SCANPY интегрирует канонические методы анализа в масштабируемом виде
SCANPY интегрирует возможности анализа установленных фреймворков на основе R и предоставляет их в масштабируемой и модульной форме. В частности, SCANPY обеспечивает предварительную обработку, сравнимую с SEURAT [10] и CELL RANGER [6], визуализацию с помощью TSNE [11, 12], рисование графиков [13–15] и диффузионные карты [11, 16, 17], кластеризацию, аналогичную PHENOGRAPH. [18–20], идентификация маркерных генов для кластеров с помощью тестов дифференциальной экспрессии и псевдовременного упорядочения с помощью диффузионного псевдовремени [21], что выгодно отличается [22] от MONOCLE 2 [22] и WISHBONE [23] (рис. 1a).
[18–20], идентификация маркерных генов для кластеров с помощью тестов дифференциальной экспрессии и псевдовременного упорядочения с помощью диффузионного псевдовремени [21], что выгодно отличается [22] от MONOCLE 2 [22] и WISHBONE [23] (рис. 1a).
a Функции анализа SCANPY. Мы используем пример 68 579 мононуклеарных клеток периферической крови [6]. Мы регрессируем смешанные переменные, нормализуем и идентифицируем сильно изменчивые гены. Визуализации TSNE и графического рисунка (Фрухтерман-Рейнгольд) показывают аннотации клеточного типа, полученные путем сравнения с массовым выражением. Клетки группируются с использованием алгоритма Лувена. Ранжирование дифференциально экспрессируемых генов в кластерах идентифицирует маркерный ген MS4A1 для В-клеток в кластере 7, что согласуется с общими метками. Мы используем псевдовременное упорядочение от корневой клетки в кластере CD34+ и обнаруживаем траекторию ветвления, визуализируемую с помощью карт TSNE и диффузии. b Набор для ускорения CELL RANGER R. Рассмотрим репрезентативные шаги анализа [6]. c Визуализация и кластеризация 1,3 миллиона клеток. Данные о клетках мозга мышей E18 находятся в открытом доступе на сайте 10x Genomics. PCA = анализ главных компонент, DC = компонент диффузии
b Набор для ускорения CELL RANGER R. Рассмотрим репрезентативные шаги анализа [6]. c Визуализация и кластеризация 1,3 миллиона клеток. Данные о клетках мозга мышей E18 находятся в открытом доступе на сайте 10x Genomics. PCA = анализ главных компонент, DC = компонент диффузии
Полноразмерное изображение
SCANPY сравнивается с установленными пакетами
В подробном руководстве по кластеризации 2700 мононуклеарных клеток периферической крови (PBMC), адаптированном из одного из руководств SEURAT (http://satijalab.org/seurat/pbmc3k_tutorial.html) [2], выполняются все шаги, начиная с необработанных данных подсчета и заканчивая идентификацией типов клеток, обеспечивая ускорение от 5 до 90 раз на каждом этапе (https://github.com/theislab/scanpy_usage/tree/master/170505_seurat). Сравнивая с более оптимизированным во время выполнения комплектом CELL RANGER R [6], мы демонстрируем ускорение от 5 до 16 раз для набора данных из 68 579 PBMC (рис. 1a,b, https://github.com/theislab/scanpy_usage). /дерево/мастер/170503_zheng17) [6]. Кроме того, мы демонстрируем возможность анализа 1,3 миллиона ячеек без подвыборки за несколько часов вычислительного времени на восьми ядрах небольшого вычислительного сервера (рис. 1c, https://github.com/theislab/scanpy_usage/tree/master/170522_visualizing_one_million_cells). ). Таким образом, SCANPY предоставляет инструменты с ускорением, которые позволяют анализировать наборы данных с более чем одним миллионом ячеек и интерактивный анализ со временем выполнения порядка секунд для примерно 100 000 ячеек.
1a,b, https://github.com/theislab/scanpy_usage). /дерево/мастер/170503_zheng17) [6]. Кроме того, мы демонстрируем возможность анализа 1,3 миллиона ячеек без подвыборки за несколько часов вычислительного времени на восьми ядрах небольшого вычислительного сервера (рис. 1c, https://github.com/theislab/scanpy_usage/tree/master/170522_visualizing_one_million_cells). ). Таким образом, SCANPY предоставляет инструменты с ускорением, которые позволяют анализировать наборы данных с более чем одним миллионом ячеек и интерактивный анализ со временем выполнения порядка секунд для примерно 100 000 ячеек.
В дополнение к упомянутым стандартным подходам анализа на основе кластеризации мы демонстрируем реконструкцию ветвящихся процессов развития с помощью диффузионного псевдовремени [21], как и в оригинальной статье (https://github.com/theislab/scanpy_usage/tree/master/ 170502_haghverdi16), моделирование одиночных клеток с помощью курируемых в литературе сетей регуляции генов на основе идей [24] (https://github. com/theislab/scanpy_usage/tree/master/170430_krumsiek11), а также анализ методов глубокого обучения результаты для данных визуализации одиночных клеток [25] (https://github.com/theislab/scanpy_usage/tree/master/170529_картинки).
com/theislab/scanpy_usage/tree/master/170430_krumsiek11), а также анализ методов глубокого обучения результаты для данных визуализации одиночных клеток [25] (https://github.com/theislab/scanpy_usage/tree/master/170529_картинки).
SCANPY представляет эффективные варианты модульной реализации
В SCANPY мы вводим класс ANNDATA — с соответствующим пакетом ANNDATA — который хранит матрицу данных с наиболее общими возможными аннотациями: аннотации наблюдений (выборки, ячейки) и переменных (признаки, гены) и неструктурированные аннотации. Поскольку SCANPY построен вокруг этого класса, в набор инструментов легко добавить новые функции. Все инструменты статистики и машинного обучения извлекают информацию из матрицы данных, которую можно добавить к объекту ANNDATA, не затрагивая структуру ANNDATA. ANNDATA похож на EXPRESSIONSET [26] в R, но поддерживает разреженные данные и позволяет создавать резервные копии объектов ANNDATA на основе HDF5 на диске — формат, не зависящий от платформы, фреймворка и языка. Это позволяет работать с объектом ANNDATA, не загружая его полностью в память — функциональность предлагается в резервном режиме ANNDATA, а не в режиме памяти. Чтобы упростить конвейеры с эффективным использованием памяти, функции SCANPY по умолчанию работают на месте, но допускают дополнительное неразрушающее преобразование объектов. Конвейеры, написанные таким образом, затем также могут быть запущены в резервном режиме для использования формулировок алгоритмов онлайн-обучения. Почти все инструменты SCANPY распараллелены.
Это позволяет работать с объектом ANNDATA, не загружая его полностью в память — функциональность предлагается в резервном режиме ANNDATA, а не в режиме памяти. Чтобы упростить конвейеры с эффективным использованием памяти, функции SCANPY по умолчанию работают на месте, но допускают дополнительное неразрушающее преобразование объектов. Конвейеры, написанные таким образом, затем также могут быть запущены в резервном режиме для использования формулировок алгоритмов онлайн-обучения. Почти все инструменты SCANPY распараллелены.
SCANPY представляет класс для представления графа отношений соседства между точками данных. Вычисление отношений соседства происходит намного быстрее, чем в популярном справочном пакете [27]. Это достигается путем агрегирования строк (наблюдений) в матрице данных в подматрицы и вычисления расстояний для каждой подматрицы с использованием быстрого параллелизованного матричного умножения. Кроме того, класс предоставляет несколько функций для вычисления метрик на основе случайного блуждания, которые недоступны в других графических программах [14, 28, 29]. ]. Как правило, инструменты SCANPY повторно используют однажды вычисленное единое графическое представление данных и, следовательно, избегают использования различных, потенциально противоречивых и ресурсоемких представлений данных.
]. Как правило, инструменты SCANPY повторно используют однажды вычисленное единое графическое представление данных и, следовательно, избегают использования различных, потенциально противоречивых и ресурсоемких представлений данных.
Выводы
Масштабируемость SCANPY напрямую удовлетворяет постоянно растущую потребность в агрегировании все больших и больших наборов данных [30] в различных экспериментальных установках, например, в рамках таких задач, как Атлас клеток человека [31]. Более того, будучи реализованным по модульному принципу, SCANPY может легко развиваться и поддерживаться сообществом. Передача результатов, полученных с помощью различных инструментов, используемых в сообществе, проста, поскольку форматы и объекты хранения данных SCANPY не зависят от языка и являются кросс-платформенными. SCANPY хорошо интегрируется в существующую экосистему Python, в которой еще нет сопоставимого инструментария.
Во время пересмотра этой статьи был предложен формат файла ткацкого станка (https://github. com/linnarsson-lab/loompy) для хранения аннотированных данных на основе HDF5. В рамках совместных усилий по облегчению обмена данными между различными лабораториями ANNDATA теперь поддерживает импорт и экспорт в ткацкий станок (https://github.com/linnarsson-lab/loompy). В этом контексте мы признательны за обсуждения с С. Линнарсоном, которые побудили нас расширить ранее статическую поддержку ANNDATA до динамической поддержки HDF5. Непосредственно перед отправкой этой рукописи библиотека C++, которая обеспечивает простое взаимодействие матриц на основе HDF5 в R, была доступна в виде препринта [32].
com/linnarsson-lab/loompy) для хранения аннотированных данных на основе HDF5. В рамках совместных усилий по облегчению обмена данными между различными лабораториями ANNDATA теперь поддерживает импорт и экспорт в ткацкий станок (https://github.com/linnarsson-lab/loompy). В этом контексте мы признательны за обсуждения с С. Линнарсоном, которые побудили нас расширить ранее статическую поддержку ANNDATA до динамической поддержки HDF5. Непосредственно перед отправкой этой рукописи библиотека C++, которая обеспечивает простое взаимодействие матриц на основе HDF5 в R, была доступна в виде препринта [32].
Методы
Технологические основы SCANPY
Ядро SCANPY основано на NUMPY [33], SCIPY [34], MATPLOTLIB [35], PANDAS [36] и H5PY [37]. Части инструментария основаны на SCIKIT-LEARN [27], STATSMODELS [38], SEABORN [39], NETWORKX [28], IGRAPH [14], пакете TSNE [40] и пакете кластеризации Louvain [41]. . Класс ANNDATA, доступный в пакете ANNDATA, использует только NUMPY, SCIPY, PANDAS и H5PY.
Реализация SCANPY на основе Python позволяет легко взаимодействовать с передовыми пакетами машинного обучения, такими как TENSORFLOW [9] для глубокого обучения [42], LIMIX для линейных смешанных моделей [43] и GPY/GPFLOW для гауссовских процессов [44, 45]. Тем не менее, мы отмечаем, что экосистема Python имеет меньше возможностей для классического статистического анализа по сравнению с R.
Сравнение с существующими пакетами Python для анализа отдельных клеток /scLVM) [46, 47], который использует модели латентных переменных гауссовского процесса для вывода скрытых источников вариации, существуют, среди прочего, фреймворки визуализации FASTPROJECT (https://github.com/YosefLab/FastProject) [48], АКЦИНА (http://www.cellaccense.com/) [49] и SPRING (https://github.com/AllonKleinLab/SPRING) [15] — последний использует пакет JavaScript (http://d3js.org D3.js для фактической визуализации и Python только для препроцессинга — траектория инструмент логического вывода SCIMITAR (https://github.
 com/dimenwarper/scimitar), инструмент кластеризации PHENOGRAPH (https://github.com/jacoblevine/PhenoGraph) [19], инструмент планирования экспериментов с одной ячейкой MIMOSCA (https:/ /github.com/asncd/MIMOSCA)[50], UMIS (https://github.com/vals/umis) для обработки необработанных прочитанных данных [51], инструмент вывода дерева ECLAIR (https://github.com /GGiecold/ECLAIR) [52] и фреймворк FLOTILLA (https://github.com/yeolab/flotilla), который поставляется с модулями для простой визуализации, простой кластеризации и дифференциального тестирования выражений, следовательно, только последний обеспечивает фреймворк для анализа данных, решающий более одной конкретной задачи.Однако, в отличие от SCANPY, FLOTILLA не ориентирована ни на одноячеечные, ни на крупномасштабные данные и не предоставляет графических методов, которые h являются ядром SCANPY. Кроме того, FLOTILLA построена вокруг сложного класса STUDY, который содержит данные, инструменты и функции построения графиков. SCANPY, напротив, построен на основе простого класса ANNDATA, поддерживаемого HDF5, что делает SCANPY масштабируемым и расширяемым (закон Деметры).
com/dimenwarper/scimitar), инструмент кластеризации PHENOGRAPH (https://github.com/jacoblevine/PhenoGraph) [19], инструмент планирования экспериментов с одной ячейкой MIMOSCA (https:/ /github.com/asncd/MIMOSCA)[50], UMIS (https://github.com/vals/umis) для обработки необработанных прочитанных данных [51], инструмент вывода дерева ECLAIR (https://github.com /GGiecold/ECLAIR) [52] и фреймворк FLOTILLA (https://github.com/yeolab/flotilla), который поставляется с модулями для простой визуализации, простой кластеризации и дифференциального тестирования выражений, следовательно, только последний обеспечивает фреймворк для анализа данных, решающий более одной конкретной задачи.Однако, в отличие от SCANPY, FLOTILLA не ориентирована ни на одноячеечные, ни на крупномасштабные данные и не предоставляет графических методов, которые h являются ядром SCANPY. Кроме того, FLOTILLA построена вокруг сложного класса STUDY, который содержит данные, инструменты и функции построения графиков. SCANPY, напротив, построен на основе простого класса ANNDATA, поддерживаемого HDF5, что делает SCANPY масштабируемым и расширяемым (закон Деметры).
Доступность и требования
Открытый исходный код SCANPY и ANNDATA поддерживается на GITHUB (https://github.com/theislab/scanpy, https://github.com/theislab/anndata) и публикуется под лицензией BSD3.
SCANPY и ANNDATA выпускаются через индекс пакетов Python: https://pypi.python.org/pypi/scanpy и https://pypi.python.org/pypi/anndata.
Демонстрации и тесты, обсуждаемые в основном тексте, хранятся по адресу https://github.com/theislab/scanpy_usage и обобщаются здесь:
Наборы данных, используемые в демонстрациях и тестах, представляют собой три набора данных от 10x Genomics.
Язык программирования: Python.
Операционная система: Linux, Mac OS и Windows. Нац биотехнолог. 2016; 34:1145–60.
Нац биотехнолог. 2016; 34:1145–60.
Артикул КАС пабмед ПабМед Центральный Google Scholar
Ссылки на скачивание
Благодарности
Мы благодарим авторов Seurat, Cell Ranger и spring за то, что они поделились своими замечательными руководствами. Мы благодарны Стену Линнарсону за обсуждение поддержки HDF5 данных на диске. Мы благодарим С. Тритчлера, Л. Саймона, Д. С. Фишера и М. Бюттнера за комментарии к программному пакету. Мы благодарим М. Лотфоллахи за кластеризацию набора данных из 1,3 миллиона ячеек и Н. К. Члиса за настройку инструкций по установке для Windows.
Финансирование
FAW признательна за поддержку Программы постдоков Гельмгольца, Инициативы и Сетевого фонда Ассоциации Гельмгольца. FJT выражает благодарность Немецкому исследовательскому фонду (DFG) в рамках Совместного исследовательского центра 1243, подпроект A17.
Информация об авторе
Авторы и организации
Автора. публикации
Вы также можете искать этого автора в PubMed Google Scholar
Contributions
Компания FAW задумала проект и разработала программное обеспечение. PA участвовала в разработке программного обеспечения, в основном в отношении архитектуры и удобства обслуживания. FJT руководил проектом и помогал интерпретировать и представлять результаты. FAW написал рукопись с помощью PA и FJT. Все авторы прочитали и одобрили окончательный вариант рукописи.
Авторы переписки
Переписка с Ф. Александр Вольф или Фабиан Дж. Тайс.
Декларация этики
Утверждение этики и согласие на участие
Утверждение этики не применимо к данному исследованию.
Конкурирующие интересы
Ни один из авторов не заявляет о конкурирующих интересах.
Примечание издателя
Springer Nature остается нейтральной в отношении юрисдикционных претензий в опубликованных картах и институциональной принадлежности.


 При этом измеряется жизненная емкость легких, минутный объем дыхания, скорость движения воздуха по различным отделам дыхательных путей.
При этом измеряется жизненная емкость легких, минутный объем дыхания, скорость движения воздуха по различным отделам дыхательных путей. 0004
0004 37 : 1
37 : 1 обращений
обращений 1c): https://github.com/theislab/scanpy_usage/tree/master/170522_visualizing_one_million_cells.
1c): https://github.com/theislab/scanpy_usage/tree/master/170522_visualizing_one_million_cells. MAST: гибкая статистическая основа для оценки транскрипционных изменений и характеристики гетерогенности в данных секвенирования одноклеточной РНК. Геном биол. 2015 г.; 16:278.
MAST: гибкая статистическая основа для оценки транскрипционных изменений и характеристики гетерогенности в данных секвенирования одноклеточной РНК. Геном биол. 2015 г.; 16:278. F1000Исследование. 2016; 5:2122.
F1000Исследование. 2016; 5:2122. ViSNE позволяет визуализировать высокоразмерные данные по отдельным клеткам и выявляет фенотипическую гетерогенность лейкемии. Нац биотехнолог. 2013; 31: 545–52.
ViSNE позволяет визуализировать высокоразмерные данные по отдельным клеткам и выявляет фенотипическую гетерогенность лейкемии. Нац биотехнолог. 2013; 31: 545–52. Дж. Карты диффузии для многомерного одноклеточного анализа данных дифференцировки. Биоинформатика. 2015 г.; 31:2989–98.
Дж. Карты диффузии для многомерного одноклеточного анализа данных дифференцировки. Биоинформатика. 2015 г.; 31:2989–98. Идентификация типов клеток по одноклеточным транскриптомам с использованием нового метода кластеризации. Биоинформатика. 2015 г.; 31:1974–80.
Идентификация типов клеток по одноклеточным транскриптомам с использованием нового метода кластеризации. Биоинформатика. 2015 г.; 31:1974–80. Преобразование булевых моделей в непрерывные модели: методология и применение к передаче сигналов Т-клеточного рецептора. BMC Сист Биол. 2009 г.; 3:98.
Преобразование булевых моделей в непрерывные модели: методология и применение к передаче сигналов Т-клеточного рецептора. BMC Сист Биол. 2009 г.; 3:98. А., Сварт П.Дж. Изучение структуры, динамики и функций сети с помощью networkx. В: Материалы 7-й конференции Python в науке (SciPy2008). Пасадена: 2008. с. 11–15.
А., Сварт П.Дж. Изучение структуры, динамики и функций сети с помощью networkx. В: Материалы 7-й конференции Python в науке (SciPy2008). Пасадена: 2008. с. 11–15. bioRxiv. 2017. https://doi.org/10.1101/167445.
bioRxiv. 2017. https://doi.org/10.1101/167445.
 In: van der Walt S, Millman J, (eds).Limix: генетический анализ множественных признаков; 2014 г. https://doi.org/10.1101/003905, http://conference.scipy.org/proceedings/scipy2010/mckinney.html. bioRxiv.
In: van der Walt S, Millman J, (eds).Limix: генетический анализ множественных признаков; 2014 г. https://doi.org/10.1101/003905, http://conference.scipy.org/proceedings/scipy2010/mckinney.html. bioRxiv. f-scLVM: масштабируемый и универсальный факторный анализ для одноклеточной РНК-seq. Геном биол. 2017; 18:212.
f-scLVM: масштабируемый и универсальный факторный анализ для одноклеточной РНК-seq. Геном биол. 2017; 18:212. и др. Анализ мощности экспериментов по секвенированию РНК в одной клетке. Нат Методы. 2017; 14:381.
и др. Анализ мощности экспериментов по секвенированию РНК в одной клетке. Нат Методы. 2017; 14:381. Theis
Theis